Visualisation des données sur MNIST¶
Nous allons utiliser des outils de visualisation qui vont vont permettre de représenter chaque donnée (par exemple une image de la base MNIST) par un point dans l’espace 2D.
Exercice 0 : Chargement des données¶
On va travailler avec la base de données image MNIST, constituée d’images de caractères manuscrits (60000 images en apprentissage, 10000 en test).
Voici un bout de code pour récupérer les données :
from keras.datasets import mnist # the data, shuffled and split between train and test sets (X_train, y_train), (X_test, y_test) = mnist.load_data() X_train = X_train.reshape(60000, 784) X_test = X_test.reshape(10000, 784) X_train = X_train.astype('float32') X_test = X_test.astype('float32') X_train /= 255 X_test /= 255 print(X_train.shape[0], 'train samples') print(X_test.shape[0], 'test samples')
- On va commencer par afficher les 200 premières images de la base d’apprentissage.
Écrire un script
exo0.pyqui va récupérer les données avec le code précédentCompléter
exo0.pypour permettre l’affichage demandé en utilisant le code suivant :
import matplotlib.pyplot as plt plt.figure(figsize=(7.195, 3.841), dpi=100) for i in range(200): plt.subplot(10,20,i+1) plt.imshow(X_train[i,:].reshape([28,28]), cmap='gray') plt.axis('off') plt.show()
Le script exo0.py doit produire l’affichage ci-dessous :

Question :
Quel est l’espace dans lequel se trouvent les images ? Quel est sa taille ?
Exercice 1 : Visualisation avec t-SNE¶
La méthode t-Distributed Stochastic Neighbor Embedding (t-SNE) [vdMH08] est une réduction de dimension non linéaire, dont l’objectif est d’assurer que des points proches dans l’espace de départ aient des position proches dans l’espace (2D) projeté. Dit autrement, la mesure de distance entre points dans l’espace 2D doit refléter la mesure de distance dans l’espace initial.
On va appliquer la méthode t-SNE sur les données brutes de la base de test de MNIST en utilisant la classe TSNE
du module sklearn.manifold : http://scikit-learn.org/stable/modules/generated/sklearn.manifold.TSNE.html
Créer un script exo1.py dont l’objectif va être d’effectuer une réduction de dimension en 2D des données de la base de test de MNIST en utilisant la méthode t-SNE. On aura besoin des modules suivants qu’on pourra importer en début de script :
import matplotlib as mpl
import matplotlib.pyplot as plt
import matplotlib.cm as cm
import numpy as np
from scipy.spatial import ConvexHull
from sklearn.mixture import GaussianMixture
from scipy import linalg
from sklearn.neighbors import NearestNeighbors
from sklearn.manifold import TSNE
Créer une instance de type
TSNE. N.B : on choisira 2 composantes et les paramètres suivants :init='pca'(réduire la dimension préalablement avec une ACP),perplexity=30(lié au nombre de voisins dans le calcul des distances),verbose=2(pour l’affichage lors de l’apprentissage).Appliquer la transformation pour obtenir les données projetées en 2D (fonction
fit_transform). N.B : essayer tout d’abord avec un sous ensemble de la base (e.g. 1000 exemples) pour tester l’algorithme, l’apprentissage avec l’ensemble de la base de test pouvant être long.
Métrique de séparation des classes¶
On va maintenant compléter le script exo1.py précédent afin de visualiser l’ensemble des points projetés en 2D, et de définir
des critères pour analyser la séparabilité des classes dans l’espace projeté.
1. Calcul de l’enveloppe convexe des points projetés pour chacune des classe classe.
On utilisera pour cela la la classe ConvexHull du module scipy.spatial https://docs.scipy.org/doc/scipy/reference/generated/scipy.spatial.ConvexHull.html.
Sur la base MNIST, on pourra donc utiliser le code suivant pour calculer les enveloppes convexes des points pour les 10 classes :
def convexHulls(points, labels):
# computing convex hulls for a set of points with asscoiated labels
convex_hulls = []
for i in range(10):
convex_hulls.append(ConvexHull(points[labels==i,:]))
return convex_hulls
# Function Call
convex_hulls= convexHulls(x2d, labels)
où points (resp. labels) dans la méthode convexHulls(points, labels) correspond aux images projetées dans le plan 2D avec la méthode t-SNE de l’exercice 1 (resp. aux labels, i.e. classes, des images).
2. Calcul de l’ellipse de meilleure approximation des points.
On utilisera pour cela la classe GaussianMixture du module sklearn.mixture http://scikit-learn.org/stable/modules/generated/sklearn.mixture.GaussianMixture.html#sklearn.mixture.GaussianMixture.
On pourra donc utiliser le code suivant pour calculer les ellipses de meilleure approximation pour les 10 classes :
def best_ellipses(points, labels):
# computing best fiiting ellipse for a set of points with asscoiated labels
gaussians = []
for i in range(10):
gaussians.append(GaussianMixture(n_components=1, covariance_type='full').fit(points[labels==i, :]))
return gaussians
# Function Call
ellipses = best_ellipses(x2d, labels)
3. Calcul du « Neighborhood Hit » (NH) [PNML08].
Pour chaque point, la métrique NH consiste à calculer, pour les k plus proches voisins (k-nn) de ce point, le taux des voisins qui sont de la même classe que le point considéré. La métrique NH est ensuite moyennée sur l’ensemble de la base.
Le code suivant permet de calculer la métrique NH, en utilisant la classe NearestNeighbors du module sklearn.neighbors :
def neighboring_hit(points, labels):
k = 6
nbrs = NearestNeighbors(n_neighbors=k+1, algorithm='ball_tree').fit(points)
distances, indices = nbrs.kneighbors(points)
txs = 0.0
txsc = [0.0, 0.0, 0.0, 0.0, 0.0, 0.0, 0.0, 0.0, 0.0, 0.0]
nppts = [0.0, 0.0, 0.0, 0.0, 0.0, 0.0, 0.0, 0.0, 0.0, 0.0]
for i in range(len(points)):
tx = 0.0
for j in range(1,k+1):
if (labels[indices[i,j]]== labels[i]):
tx += 1
tx /= k
txsc[labels[i]] += tx
nppts[labels[i]] += 1
txs += tx
for i in range(10):
txsc[i] /= nppts[i]
return txs / len(points)
# Function Call
nh = neighboring_hit(x2d, labels)
Question :
En quoi les trois métriques ci-dessus sont-elles liées au problème de la séparabilité des classes ? Qu’est-ce qui les diffère ?
Compléter le script exo4.py pour calculer les différentes métriques.
Vous pouvez ensuite utiliser la fonction
visualizationsuivante pour afficher les points ainsi que leur labels, et de visualiser les trois métriques précédentes :
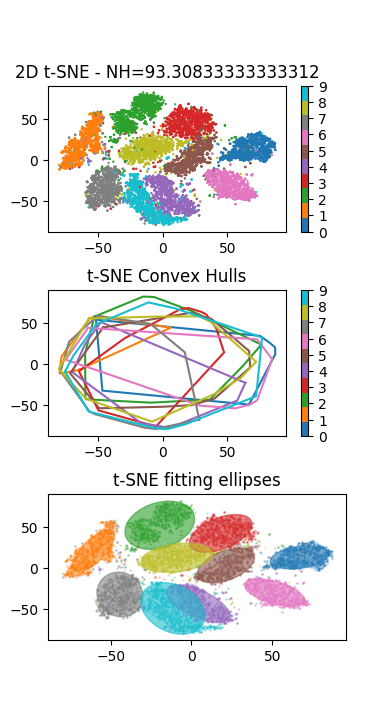
def visualization(points2D, labels, convex_hulls, ellipses ,projname, nh):
points2D_c= []
for i in range(10):
points2D_c.append(points2D[labels==i, :])
# Data Visualization
cmap =cm.tab10
plt.figure(figsize=(3.841, 7.195), dpi=100)
plt.set_cmap(cmap)
plt.subplots_adjust(hspace=0.4 )
plt.subplot(311)
plt.scatter(points2D[:,0], points2D[:,1], c=labels, s=3,edgecolors='none', cmap=cmap, alpha=1.0)
plt.colorbar(ticks=range(10))
plt.title("2D "+projname+" - NH="+str(nh*100.0))
vals = [ i/10.0 for i in range(10)]
sp2 = plt.subplot(312)
for i in range(10):
ch = np.append(convex_hulls[i].vertices,convex_hulls[i].vertices[0])
sp2.plot(points2D_c[i][ch, 0], points2D_c[i][ch, 1], '-',label='$%i$'%i, color=cmap(vals[i]))
plt.colorbar(ticks=range(10))
plt.title(projname+" Convex Hulls")
def plot_results(X, Y_, means, covariances, index, title, color):
splot = plt.subplot(3, 1, 3)
for i, (mean, covar) in enumerate(zip(means, covariances)):
v, w = linalg.eigh(covar)
v = 2. * np.sqrt(2.) * np.sqrt(v)
u = w[0] / linalg.norm(w[0])
# as the DP will not use every component it has access to
# unless it needs it, we shouldn't plot the redundant
# components.
if not np.any(Y_ == i):
continue
plt.scatter(X[Y_ == i, 0], X[Y_ == i, 1], .8, color=color, alpha = 0.2)
# Plot an ellipse to show the Gaussian component
angle = np.arctan(u[1] / u[0])
angle = 180. * angle / np.pi # convert to degrees
ell = mpl.patches.Ellipse(mean, v[0], v[1], 180. + angle, color=color)
ell.set_clip_box(splot.bbox)
ell.set_alpha(0.6)
splot.add_artist(ell)
plt.title(title)
plt.subplot(313)
for i in range(10):
plot_results(points2D[labels==i, :], ellipses[i].predict(points2D[labels==i, :]), ellipses[i].means_,
ellipses[i].covariances_, 0,projname+" fitting ellipses", cmap(vals[i]))
plt.savefig(projname+".png", dpi=100)
plt.show()
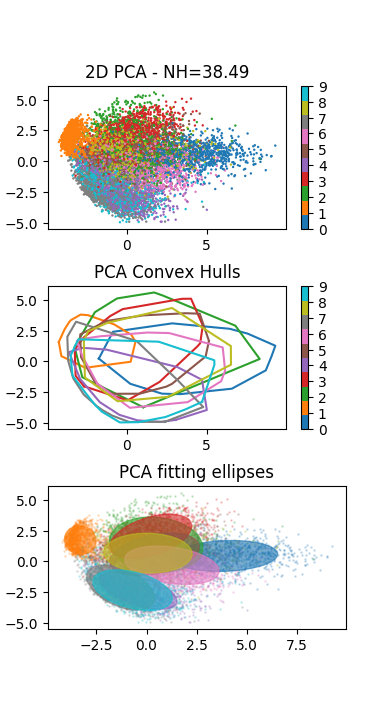
Comparer la méthode t-SNE à une Analyse en Composantes Principales (ACP) [Hot33]. On pourra utiliser la classe
PCAdu modulesklearn.decompositionhttp://scikit-learn.org/stable/modules/generated/sklearn.decomposition.PCA.html.L’application de la méthode visualisation sur les données de test de la base MNIST doit produire le résultat suivant (à gauche le résultat de la t-SNE à droite de l’ACP) :
Analyser la distribution des points et des classes : que peut-on en conclure ?
- Hot33
H. Hotelling. Analysis of a Complex of Statistical Variables Into Principal Components. Warwick & York, 1933. URL: https://books.google.fr/books?id=qJfXAAAAMAAJ.
- PNML08
Fernando Vieira Paulovich, Luis Gustavo Nonato, Rosane Minghim, and Haim Levkowitz. Least square projection: A fast high-precision multidimensional projection technique and its application to document mapping. IEEE Trans. Vis. Comput. Graph., 14(3):564–575, 2008.
- vdMH08
Laurens van der Maaten and Geoffrey E. Hinton. Visualizing high-dimensional data using t-sne. Journal of Machine Learning Research, 9:2579–2605, 2008.